svm — libsvm (C ++) всегда выводит одно и то же предсказание
Я реализовал оболочку OpenCV / C ++ для libsvm. При выполнении сетки поиска параметров SVM (ядро RBF) прогнозирование всегда возвращает тот же ярлык. Я создал искусственные наборы данных, которые имеют очень легко разделяемые данные (и попытался предсказать данные, на которых я только что обучался), но, тем не менее, он возвращает ту же самую метку.
Я использовал MATLAB-реализацию libsvm и добился высокой точности на том же наборе данных. Должно быть, я что-то делаю не так с установкой проблемы, но я много раз проходил через README и не могу найти проблему.
Вот как я настроил проблему libsvm, где data — это OpenCV Mat:
const int rowSize = data.rows;
const int colSize = data.cols;
this->_svmProblem = new svm_problem;
std::memset(this->_svmProblem,0,sizeof(svm_problem));
//dynamically allocate the X matrix...
this->_svmProblem->x = new svm_node*[rowSize];
for(int row = 0; row < rowSize; ++row)
this->_svmProblem->x[row] = new svm_node[colSize + 1];
//...and the y vector
this->_svmProblem->y = new double[rowSize];
this->_svmProblem->l = rowSize;
for(int row = 0; row < rowSize; ++row)
{
for(int col = 0; col < colSize; ++col)
{
//set the index and the value. indexing starts at 1.
this->_svmProblem->x[row][col].index = col + 1;
double tempVal = (double)data.at<float>(row,col);
this->_svmProblem->x[row][col].value = tempVal;
}
this->_svmProblem->x[row][colSize].index = -1;
this->_svmProblem->x[row][colSize].value = 0;
//add the label to the y array, and feature vector to X matrix
double tempVal = (double)labels.at<float>(row);
this->_svmProblem->y[row] = tempVal;
}}/*createProblem()*/
Вот как я настраиваю параметры, где svmParams — это моя собственная структура для C / Gamma и такие:
this->_svmParameter = new svm_parameter;
std::memset(this->_svmParameter,0,sizeof(svm_parameter));
this->_svmParameter->svm_type = svmParams.svmType;
this->_svmParameter->kernel_type = svmParams.kernalType;
this->_svmParameter->C = svmParams.C;
this->_svmParameter->gamma = svmParams.gamma;
this->_svmParameter->nr_weight = 0;
this->_svmParameter->eps = 0.001;
this->_svmParameter->degree = 1;
this->_svmParameter->shrinking = 0;
this->_svmParameter->probability = 1;
this->_svmParameter->cache_size = 100;
Я использую предоставленную функцию проверки параметров / проблем, и ошибки не возвращаются.
Я тогда тренируюсь как таковой:
this->_svmModel = svm_train(this->_svmProblem, this->_svmParameter);
А потом предсказать так:
float pred = (float)svm_predict(this->_svmModel, x[i]);
Если бы кто-то мог указать, где я иду не так, я был бы очень признателен. Спасибо!
РЕДАКТИРОВАТЬ:
Используя этот код, я распечатал содержание задачи
for(int i = 0; i < rowSize; ++i)
{
std::cout << "[";
for(int j = 0; j < colSize + 1; ++j)
{
std::cout << " (" << this->_svmProblem->x[i][j].index << ", " << this->_svmProblem->x[i][j].value << ")";
}
std::cout << "]" << " <" << this->_svmProblem->y[i] << ">" << std::endl;
}
Вот вывод:
[ (1, -1) (2, 0) (-1, 0)] <1>
[ (1, -0.92394) (2, 0) (-1, 0)] <1>
[ (1, -0.7532) (2, 0) (-1, 0)] <1>
[ (1, -0.75977) (2, 0) (-1, 0)] <1>
[ (1, -0.75337) (2, 0) (-1, 0)] <1>
[ (1, -0.76299) (2, 0) (-1, 0)] <1>
[ (1, -0.76527) (2, 0) (-1, 0)] <1>
[ (1, -0.74631) (2, 0) (-1, 0)] <1>
[ (1, -0.85153) (2, 0) (-1, 0)] <1>
[ (1, -0.72436) (2, 0) (-1, 0)] <1>
[ (1, -0.76485) (2, 0) (-1, 0)] <1>
[ (1, -0.72936) (2, 0) (-1, 0)] <1>
[ (1, -0.94004) (2, 0) (-1, 0)] <1>
[ (1, -0.92756) (2, 0) (-1, 0)] <1>
[ (1, -0.9688) (2, 0) (-1, 0)] <1>
[ (1, 0.05193) (2, 0) (-1, 0)] <3>
[ (1, -0.048488) (2, 0) (-1, 0)] <3>
[ (1, 0.070436) (2, 0) (-1, 0)] <3>
[ (1, 0.15191) (2, 0) (-1, 0)] <3>
[ (1, -0.07331) (2, 0) (-1, 0)] <3>
[ (1, 0.019786) (2, 0) (-1, 0)] <3>
[ (1, -0.072793) (2, 0) (-1, 0)] <3>
[ (1, 0.16157) (2, 0) (-1, 0)] <3>
[ (1, -0.057188) (2, 0) (-1, 0)] <3>
[ (1, -0.11187) (2, 0) (-1, 0)] <3>
[ (1, 0.15886) (2, 0) (-1, 0)] <3>
[ (1, -0.0701) (2, 0) (-1, 0)] <3>
[ (1, -0.17816) (2, 0) (-1, 0)] <3>
[ (1, 0.12305) (2, 0) (-1, 0)] <3>
[ (1, 0.058615) (2, 0) (-1, 0)] <3>
[ (1, 0.80203) (2, 0) (-1, 0)] <1>
[ (1, 0.734) (2, 0) (-1, 0)] <1>
[ (1, 0.9072) (2, 0) (-1, 0)] <1>
[ (1, 0.88061) (2, 0) (-1, 0)] <1>
[ (1, 0.83903) (2, 0) (-1, 0)] <1>
[ (1, 0.86604) (2, 0) (-1, 0)] <1>
[ (1, 1) (2, 0) (-1, 0)] <1>
[ (1, 0.77988) (2, 0) (-1, 0)] <1>
[ (1, 0.8578) (2, 0) (-1, 0)] <1>
[ (1, 0.79559) (2, 0) (-1, 0)] <1>
[ (1, 0.99545) (2, 0) (-1, 0)] <1>
[ (1, 0.78376) (2, 0) (-1, 0)] <1>
[ (1, 0.72177) (2, 0) (-1, 0)] <1>
[ (1, 0.72619) (2, 0) (-1, 0)] <1>
[ (1, 0.80149) (2, 0) (-1, 0)] <1>
[ (1, 0.092327) (2, -1) (-1, 0)] <2>
[ (1, 0.019054) (2, -1) (-1, 0)] <2>
[ (1, 0.15287) (2, -1) (-1, 0)] <2>
[ (1, -0.1471) (2, -1) (-1, 0)] <2>
[ (1, -0.068182) (2, -1) (-1, 0)] <2>
[ (1, -0.094567) (2, -1) (-1, 0)] <2>
[ (1, -0.17071) (2, -1) (-1, 0)] <2>
[ (1, -0.16646) (2, -1) (-1, 0)] <2>
[ (1, -0.030421) (2, -1) (-1, 0)] <2>
[ (1, 0.094346) (2, -1) (-1, 0)] <2>
[ (1, -0.14408) (2, -1) (-1, 0)] <2>
[ (1, 0.090025) (2, -1) (-1, 0)] <2>
[ (1, 0.043706) (2, -1) (-1, 0)] <2>
[ (1, 0.15065) (2, -1) (-1, 0)] <2>
[ (1, -0.11751) (2, -1) (-1, 0)] <2>
[ (1, -0.02324) (2, 1) (-1, 0)] <2>
[ (1, 0.0080356) (2, 1) (-1, 0)] <2>
[ (1, -0.17752) (2, 1) (-1, 0)] <2>
[ (1, 0.011135) (2, 1) (-1, 0)] <2>
[ (1, -0.029063) (2, 1) (-1, 0)] <2>
[ (1, 0.15398) (2, 1) (-1, 0)] <2>
[ (1, 0.097746) (2, 1) (-1, 0)] <2>
[ (1, 0.01018) (2, 1) (-1, 0)] <2>
[ (1, 0.015592) (2, 1) (-1, 0)] <2>
[ (1, -0.062793) (2, 1) (-1, 0)] <2>
[ (1, 0.014444) (2, 1) (-1, 0)] <2>
[ (1, -0.1205) (2, 1) (-1, 0)] <2>
[ (1, -0.18011) (2, 1) (-1, 0)] <2>
[ (1, 0.010521) (2, 1) (-1, 0)] <2>
[ (1, 0.036914) (2, 1) (-1, 0)] <2>
Здесь данные печатаются в формате [(index, value) …] метка.
Искусственный набор данных, который я только что создал, имеет 3 класса, каждый из которых легко разделяется с помощью нелинейной границы решения. Каждая строка представляет собой вектор объектов (наблюдение) с двумя объектами (координаты x, координаты y). Libsvm просит завершить каждый вектор меткой -1, так что я делаю.
EDIT2:
Это редактирование относится к моим значениям C и Gamma, используемым для обучения, а также к масштабированию данных. Я обычно данные между 0 и 1 (как предложено здесь: http://www.csie.ntu.edu.tw/~cjlin/papers/guide/guide.pdf). Я также масштабирую этот поддельный набор данных и попробую еще раз, хотя я использовал этот же точный набор данных с реализацией libsvm в MATLAB, и он мог бы отделить эти немасштабированные данные со 100% точностью.
Для C и Gamma я также использую значения, рекомендованные в руководстве. Я создаю два вектора и использую двойной вложенный цикл, чтобы попробовать все комбинации:
std::vector<double> CList, GList;
double baseNum = 2.0;
for(double j = -5; j <= 15; j += 2) //-5 and 15
CList.push_back(pow(baseNum,j));
for(double j = -15; j <= 3; j += 2) //-15 and 3
GList.push_back(pow(baseNum,j));
И цикл выглядит так:
for(auto CIt = CList.begin(); CIt != CList.end(); ++CIt) //for all C's
{
double C = *CIt;
for(auto GIt = GList.begin(); GIt != GList.end(); ++GIt) //for all gamma's
{
double gamma = *GIt;
svmParams.svmType = C_SVC;
svmParams.kernalType = RBF;
svmParams.C = C;
svmParams.gamma = gamma;
......training code etc..........
EDIT3:
Поскольку я продолжаю ссылаться на MATLAB, я покажу разницу в точности. Вот тепловая карта точности, которую дает libsvm:
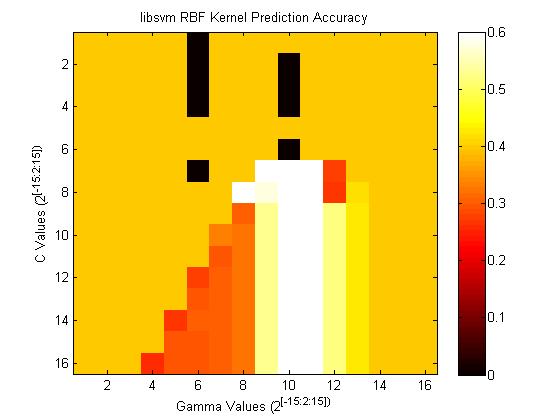
А вот карта точности, которую дает MATLAB, используя те же параметры и ту же C / Gamma grid:
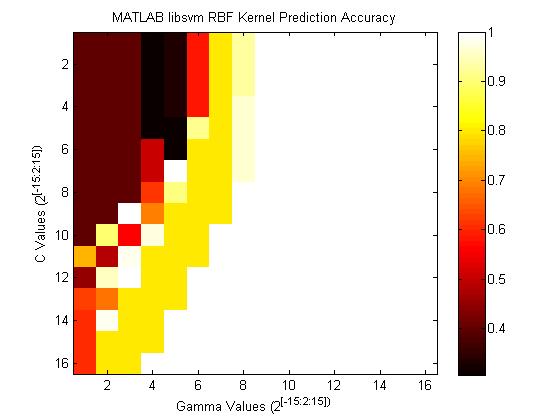
Вот код, используемый для создания списков C / Gamma, и как я тренируюсь:
CList = 2.^(-15:2:15);%(-5:2:15);
GList = 2.^(-15:2:15);%(-15:2:3);
cmd = ['-q -s 0 -t 2 -c ', num2str(C), ' -g ', num2str(gamma)];
model = ovrtrain(yTrain,xTrain,cmd);
EDIT4
В качестве проверки работоспособности я переформатировал свой поддельный масштабированный набор данных, чтобы он соответствовал набору данных, используемому терминальным API Unix / Linux libsvm. Я тренировался и предсказывал, используя C / Gamma, найденную на карте точности MATLAB. Точность прогноза составила 100%. Таким образом, я абсолютно что-то не так делаю в реализации C ++.
EDIT5
Я загрузил обученную модель из терминала Linux в мой класс оболочки C ++. Затем я попытался предсказать тот же точный набор данных, который использовался для обучения. Точность в C ++ все еще была ужасной! Однако я очень близок к тому, чтобы сузить источник проблемы. Если обе системы MATLAB / Linux согласуются с точки зрения точности 100%, и уже доказано, что модель, которую она производит, дает точность 100% для того же набора данных, на котором обучалась, и теперь мой класс-оболочка C ++ показывает низкую производительность с проверенной моделью. .. возможны три ситуации:
- Метод, который я использую для преобразования cv :: Mats в svm_node *, который требуется для прогнозирования, имеет проблему.
- В методе, который я использую для прогнозирования меток, есть проблема.
- ОБА 2 и 3!
Код, который нужно проверить, — это то, как я создаю svm_node. Здесь это снова:
svm_node** LibSVM::createNode(INPUT const cv::Mat& data)
{
const int rowSize = data.rows;
const int colSize = data.cols;
//dynamically allocate the X matrix...
svm_node** x = new svm_node*[rowSize];
if(x == NULL)
throw MLInterfaceException("Could not allocate SVM Node Array.");
for(int row = 0; row < rowSize; ++row)
{
x[row] = new svm_node[colSize + 1]; //+1 here for the index-terminating -1
if(x[row] == NULL)
throw MLInterfaceException("Could not allocate SVM Node.");
}
for(int row = 0; row < rowSize; ++row)
{
for(int col = 0; col < colSize; ++col)
{
double tempVal = data.at<double>(row,col);
x[row][col].value = tempVal;
}
x[row][colSize].index = -1;
x[row][colSize].value = 0;
}
return x;
} /*createNode()*/
И прогноз:
cv::Mat LibSVM::predict(INPUT const cv::Mat& data)
{
if(this->_svmModel == NULL)
throw MLInterfaceException("Cannot predict; no model has been trained or loaded.");
cv::Mat predMat;
//create the libsvm representation of data
svm_node** x = this->createNode(data);
//perform prediction for each feature vector
for(int i = 0; i < data.rows; ++i)
{
double pred = svm_predict(this->_svmModel, x[i]);
predMat.push_back<double>(pred);
}
//delete all rows and columns of x
for(int i = 0; i < data.rows; ++i)
delete[] x[i];
delete[] x;return predMat;
}
EDIT6:
Для тех из вас, кто настраивается дома, я обучил модель (используя оптимальную C / Gamma, найденную в MATLAB) на C ++, сохранил ее в файл, а затем попытался прогнозировать данные обучения через терминал Linux. Он набрал 100%. Что-то не так с моим прогнозом. o_0
EDIT7:
Я наконец нашел проблему. У меня была огромная помощь в обнаружении ошибок. Я распечатал содержимое двумерного массива svm_node **, используемого для прогнозирования. Это было подмножество метода createProblem (). Был фрагмент, который я не смог скопировать + вставить в новую функцию. Это был индекс данной функции; это никогда не было написано. Там должно было быть еще 1 строка:
x[row][col].index = col + 1; //indexing starts at 1
И прогноз теперь работает хорошо.
Решение
Было бы полезно увидеть значение гаммы, так как ваши данные не нормализованы, что имело бы огромное значение.
Гамма в libsvm обратно пропорциональна радиусу гиперсферы, поэтому, если эти сферы слишком малы по отношению к входному диапазону, все будет активировано всегда, и тогда модель будет всегда выводить одно и то же значение.
Итак, две рекомендации: 1) масштабируйте входные значения до диапазона [-1,1]. 2) Играйте со значениями гаммы.
Другие решения
Других решений пока нет …
